Als je in Google Afbeeldingen ‘Jaguar’ intikt, krijg je plaatjes van een zwarte panter, een gevlekte katachtige, een auto, een vliegtuig en een boot. Wie afbeeldingen gaat digitaliseren, loopt tegen dat probleem aan: hoe weet een computer wat er op zijn digitale bestanden te zien is? En meer nog: wat een gebruiker eigenlijk zoekt? Fons Verbeek van het Leiden Institute of Advanced Computer Science van de Universiteit Leiden zocht – en vond – samenwerkend binnen het Cyttron II-consortium een oplossing.
Cyttron I
2006-2010

Allemaal Jaguars…
Samengesteld beeld, credits met de klok mee; Jaguar; Wikimedia Commons, SAC Scott Lewis/MOD via Open Government Licence v1.0; Wikimedia Commons, Adrian Pingstone via CC0; en Wikimedia Commons, Colin M.L. Burnett via CC BY-SA 3.0Het idee achter het project is dat een medisch onderzoeker meer verbanden kan leggen door beelden op verschillend inzoomniveau en biochemische informatie aan elkaar te koppelen. Sterker nog: dat een database door slimme algoritmes de onderzoeker wijst op verbanden die hij zelf nog niet gezien had. Maar dat idee is sneller geschetst dan ingevuld: een celorganel is totaal onzichtbaar op een röntgenfoto, je ziet hooguit contouren op een lichtmicroscoop, en pas bij een fluorescentie- en confocale microscoop wordt het wat.
Een elektronenmicroscoop is dan vaak alweer zo sterk vergrotend, dat je naar een onderdeel van een organel zit te kijken. En dat is niet het enige probleem. ‘Zwart’ betekent in het ene geval iets compleet anders dan in het andere. ‘Paars’ hangt af van welke kleuring is gebruikt. En sommige microscopen leveren alleen zwart-witbeelden. We proberen aanvankelijk orde te scheppen met technieken die lijken op technieken die worden gebruikt bij gezichtsherkenning op foto’s. Alleen, dat loopt compleet vast. Natuurlijk: als iemand met een doos vol glaasjes met coupes van darmweefsel met dezelfde kleuring aankomt, kan een computer na het derde glaasje echt wel slimme suggesties doen. Het gaat mank als je met een nieuwe doos glaasjes met een net iets andere kleuring binnenkomt. Het moet dus anders. Weg met de pixels! Taal moet het zijn.
Uiteindelijk moet een computer toch weten wat die vage opeenhoping paars linksboven in beeld eigenlijk voorstelt, zodat hij een onderzoeker kan wijzen op de aanwezigheid van celwoekering. We besluiten te gaan experimenteren met ontologische beschrijving: een set beschrijvende termen waarbij niet alleen gedefinieerd is wat die termen betekenen, maar ook beschreven staat hoe die betekenissen relaties hebben. Ontologische beschrijvingen bestaan al heel lang. Zo weten we bijvoorbeeld dat ‘bladeren’ een vast onderdeel is van de verzameling ‘loofbomen’, categorie ‘onderdelen’. ‘Eik’, ‘iep’ en ‘es’ zijn onderdelen van diezelfde verzameling, maar dan in de categorie ‘soorten’. Ontologieën hebben clusters, verzamelingen en een hiërarchische structuur.
Cyttron II
2011-2012
Niet de beelden zelf, maar de tags die aan de beelden zijn gehangen zijn dus leidend om de samenhang tussen die beelden te duiden. Stel, je wilt in een weefsel de ribosomen nader bestuderen, dan kun je langs de term ‘ribosomen’ helemaal inzoomen tot je op molecuulniveau naar de eiwitsynthese zit te kijken. Of uitzoomen tot de celinhoud zo vaag is dat het niet meer in onderdelen te zien is.
We bestuderen gene-ontology, een bestaande ontologie voor genetica. Al snel komen we erachter dat dat zeker bruikbaar is, maar we stuiten meteen op een netelig probleem: je moet elk beeld apart annoteren. Voor een paar beelden gaat dat nog wel, maar als je tweehonderd foto’s moet vertalen in een omvangrijke ontologie, wordt dat vervelend. Welke onderzoeker is bereid dat te doen?
Ons project verschuift van een technisch naar een menselijk probleem. Zoals gewoonlijk wordt dat de grootste uitdaging.
2013
We proberen een spelelement toe te voegen. Letterlijk: we bouwen het arcadespel Frogger in de gebruikersinterface in, waarbij het hoppen naar de overkant (wat altijd de bedoeling was van Frogger: spring met een kikker een rivier over via boomstammen) eenvoudiger wordt als termen waarop je moet ‘springen’ geannoteerd zijn.
Hiermee leren we de gebruiker dat annotatie nuttig en lonend is, en moedigen onderzoekers aan goed te annoteren, door ze na afloop te belonen met een spelletje.
2014
Een doorbraak. Niet zozeer in de techniek, maar in het denken over adequate invoer. Stel, onderzoeker Janssen logt in op de database. We weten dan dat Janssen onderzoek doet naar cellen in de hersenstam, omdat Janssen dat heeft aangegeven in zijn onderzoekersprofiel. We weten ook dat Janssen een set beelden wil annoteren die afkomstig zijn van een lichtmicroscoop. Omdat hij dat eerder heeft gedaan, of omdat hij dat aan de database laat weten.
Die twee kenmerken – Janssen, lichtmicroscoop – zijn twee enorm beperkende factoren voor de set aan ontologische elementen die Janssen überhaupt kàn annoteren. Hij ziet geen eiwitten, want die zie je niet met een lichtmicroscoop. Hij ziet ook geen spiercellen, want we weten dat Janssen hersenonderzoek doet.
De imagingtechniek en de onderzoeker – twee zaken die je in een handomdraai helder hebt – beperken wat er in beeld kan zijn. En daarmee ook de tijd die je nodig hebt voor annotatie. Andersom werkt het ook: als Janssen werkt aan stressrespons in zijn hersencoupes, dan geeft de database aanbevelingen over beelden die óók gaan over stressrespons. We ontdekken daardoor bijvoorbeeld dat de stressrespons bij een gist op moleculair niveau nauwelijks afwijkt van die van ons.
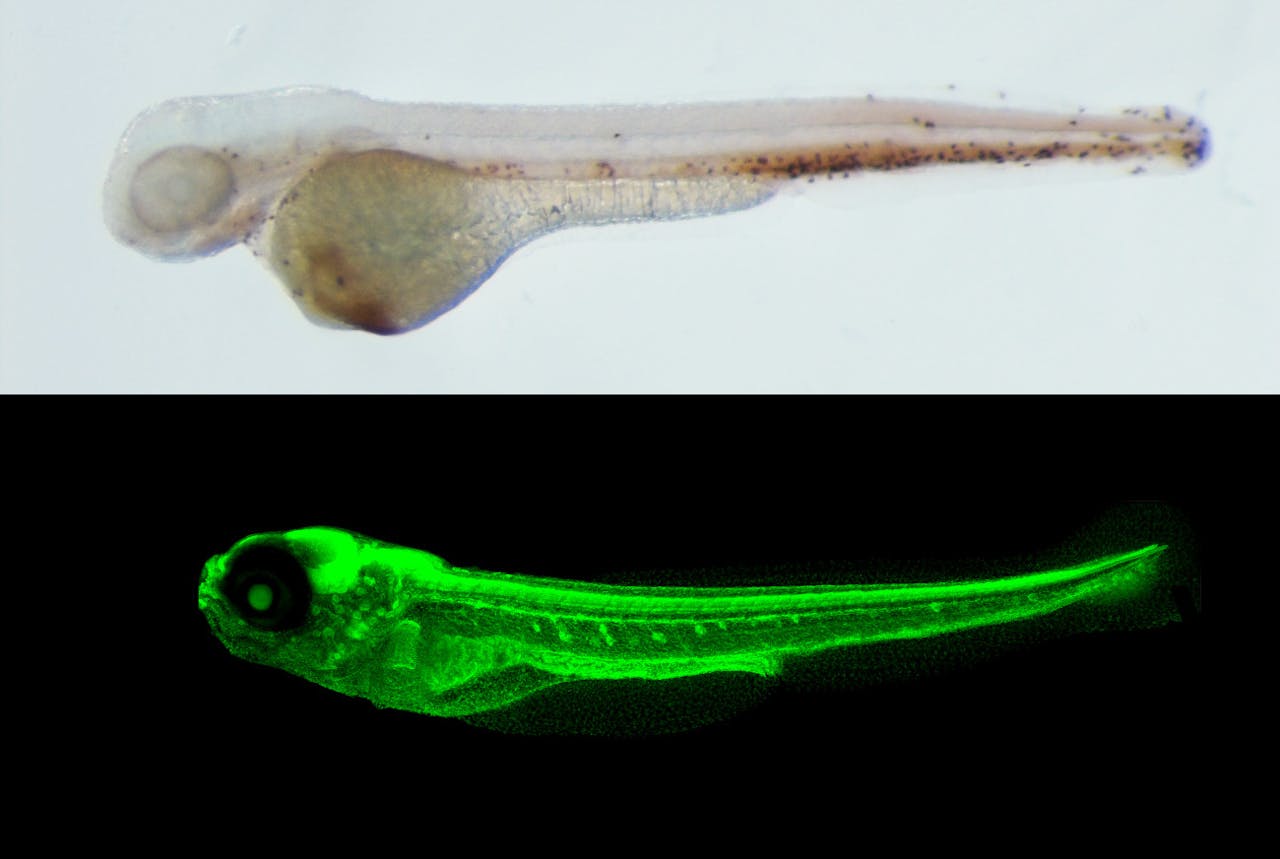
Twee keer een zebravisje: boven gefotografeerd met een gewone lichtmicroscoop en onder met een confocale microscoop.
Fons VerbeekEn nu verder…
Het klinkt allemaal mooi: een computer die verbanden legt die wetenschappers zelf over het hoofd zien. Maar de menselijke kant is een nog niet helemaal geslecht bastion. Wat we nog niet overwonnen hebben, is “vakjargon”. Als een structuurbioloog het over een “atlas” heeft, bedoelt hij iets anders dan wanneer een topograaf of een orthopeed daarover praat. We zoeken nu nog naar een manier om te voorkomen dat onderzoekers volgens hun eigen jargon gaan annoteren zonder dat het duidelijk wordt via welk jargon dat is. Het succes van Cyttron II wordt in dit opzicht bepaald door de eenduidigheid waarmee het annoteren gebeurt. Dus ook al rondt Cyttron II nu af, de klus is nog niet helemaal geklaard.

