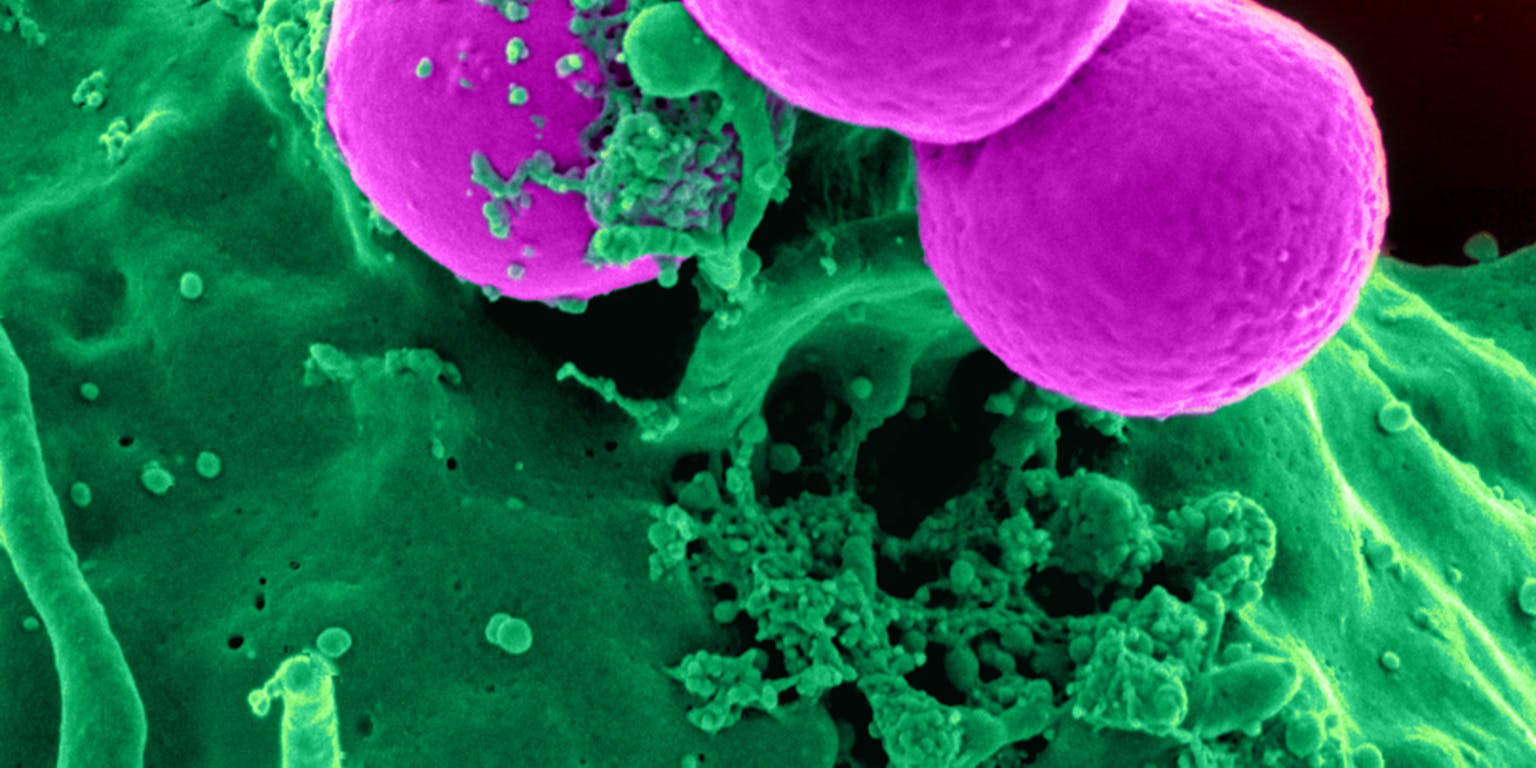
Sommige bacteriën weten het immuunsysteem heel goed te omzeilen. Andere worden grondig vernietigd. Om te begrijpen waar de verschillen zitten moeten we meer weten over hoe immuuncellen te werk gaan bij hun opruimtaken. In een publicatie in het tijdschrift Chemical Science beschrijven Leidse onderzoekers een methode om bacteriën tijdens hun afbraak zichtbaar te maken.
In een ideale wereld maken ziekteverwekkers geen schijn van kans. Het immuunsysteem herkent ze meteen en vernietigt ze voor ze kwaad kunnen doen. De realiteit is natuurlijk dat allerlei ziekmakende bacteriën, virussen en andere narigheid door de barrières heen kunnen breken, met ziekte en soms zelfs de dood tot gevolg. Promovendus Daphne van Elsland en haar collega’s van de Universiteit Leiden ontwikkelden een techniek om de bacteriën zichtbaar te maken tijdens hun confrontatie met de immuuncellen. Zo hopen ze te onthullen hoe bacteriën erin slagen het immuunsysteem om de tuin te leiden.
“Om te begrijpen hoe ons lichaam zich verdedigt tegen pathogenen (ziekteverwekkers) en waarom dat niet altijd goed gaat, moeten we veel meer weten over de interacties tussen die pathogenen en de immuuncellen”, aldus Van Elsland. Maar het bestuderen van dat proces is om meerdere redenen lastig. “Een van de problemen is dat je van bacteriën die succesvol worden vernietigd, niks terugvindt. Er zijn geen sporen waaruit je kunt afleiden hoe en waar precies ze zijn afgebroken. Een ander probleem is dat pathogenen die ons ziek maken, juist aan de immuuncellen weten te ontsnappen. Waar moet je die dan zoeken?”
Lichtgevende labels
Tijd om licht te werpen op dit proces, vond Van Elsland. Letterlijk. “We moeten die interacties zichtbaar maken zodat we bacteriën kunnen volgen gedurende de confrontatie met de immuuncellen.” Dat was nog niet mogelijk, maar door het combineren van twee bestaande technieken hebben de Leidse onderzoekers een flinke stap gezet. De eerste techniek draait om het labellen van de bacteriën. Ook dit kun je vrij letterlijk nemen: door een chemische verandering aan te brengen in de bacteriën kun je er in een volgende stap een label aanhangen. Zo’n ‘label’ is een molecuul of een chemische groep die iets speciaals kan, waardoor het zichtbaar wordt. Veel labels zijn fluorescent; ze geven licht als je ze onder een specifieke microscoop bekijkt. Zo maak je de plekken waar het label is gebonden zichtbaar.
Labelling is een veelgebruikte techniek in het onderzoek naar immuuncellen. Meestal gaat het om grote labels zoals fluorescerende eiwitten, zoals het populaire Green Fluorescent Protein (GFP), die genetisch worden ingebouwd in de bacteriën. Maar deze eiwitten zijn heel groot en verstoren daarom vaak het natuurlijke proces. Bovendien worden deze labels samen met de bacterie afgebroken, vertelt van Elsland. “Wij willen juist het natuurlijke proces bestuderen en daarom hebben we gekozen voor een chemische aanpak waarbij we een heel klein label aanbrengen. Zo klein dat het ook tijdens de afbraak intact en zichtbaar blijft. Je blijft het zien in de afgebroken onderdelen en fragmenten.” De bacteriën hebben er ogenschijnlijk geen last van “De aangepaste varianten gedragen zich niet anders dan de natuurlijke bacteriën. Ze lijken even happy.”

Bacterievoer
Normaal gesproken moet je genetische modificatie (iets veranderen in het genetisch materiaal) toepassen om een label in een bacterie te kunnen aanbrengen. Maar dat is omslachtig en het lukt vaak niet. Van Elsland gebruikt een veel eenvoudiger methode. Ze ‘voert’ een niet-natuurlijk aminozuur aan de bacteriën. Aminozuren zijn de bouwstenen van eiwitten. “Wij hebben in het groeimedium het aminozuur methionine vervangen door een variant die er heel sterk op lijkt, zodat de bacterie dit niet-natuurlijke aminozuur gewoon kan inbouwen in zijn eiwitten en normaal kan blijven functioneren. Het niet-natuurlijke aminozuur bevat de chemische groep die je later nodig hebt om je lichtgevende label aan te hangen.”
Het aanpassen van de levende bacteriën is stap een. Van Elsland legt uit wat er daarna gebeurt. “Je geeft de bacteriën aan immuuncellen en dan neem je op verschillende tijdstippen een monster. Dat snijd je in heel dunne plakjes en op die plakjes breng je het fluorescerende label aan. Dat plakt aan het aangepaste aminozuur. Vervolgens bekijk je de plakjes onder een fluorescentiemicroscoop. En dan zie je een zwart geheel met hier en daar lichtgevende vlekken.” Daar levert een mooi plaatje, maar je ziet nog steeds niet wat er gebeurt met die bacterie. Je wilt ook de directe omgeving van de bacterie zichtbaar maken.
Het bijzondere van de nieuwe methode zit in de koppeling die daarna volgt. “De doorbraak in dit project zit in het gebruik van CLEM, correlative light electron microscopy. Met deze techniek slaan we een brug tussen fluorescentiemicroscopie, waarmee we de bacterie zichtbaar maken en elektronenmicroscopie, waarmee je heel gedetailleerde de cel in beeld brengt. Je ziet allerlei onderdelen, zoals de celkern, het plasmamembraan, de mitochondriën en ga zo maar door. Door deze combinatie van labelling en twee microscopische technieken kunnen we heel goed zien waar in de immuuncellen de bacterie wordt afgebroken en waar de afbraakproducten naartoe gaan.”
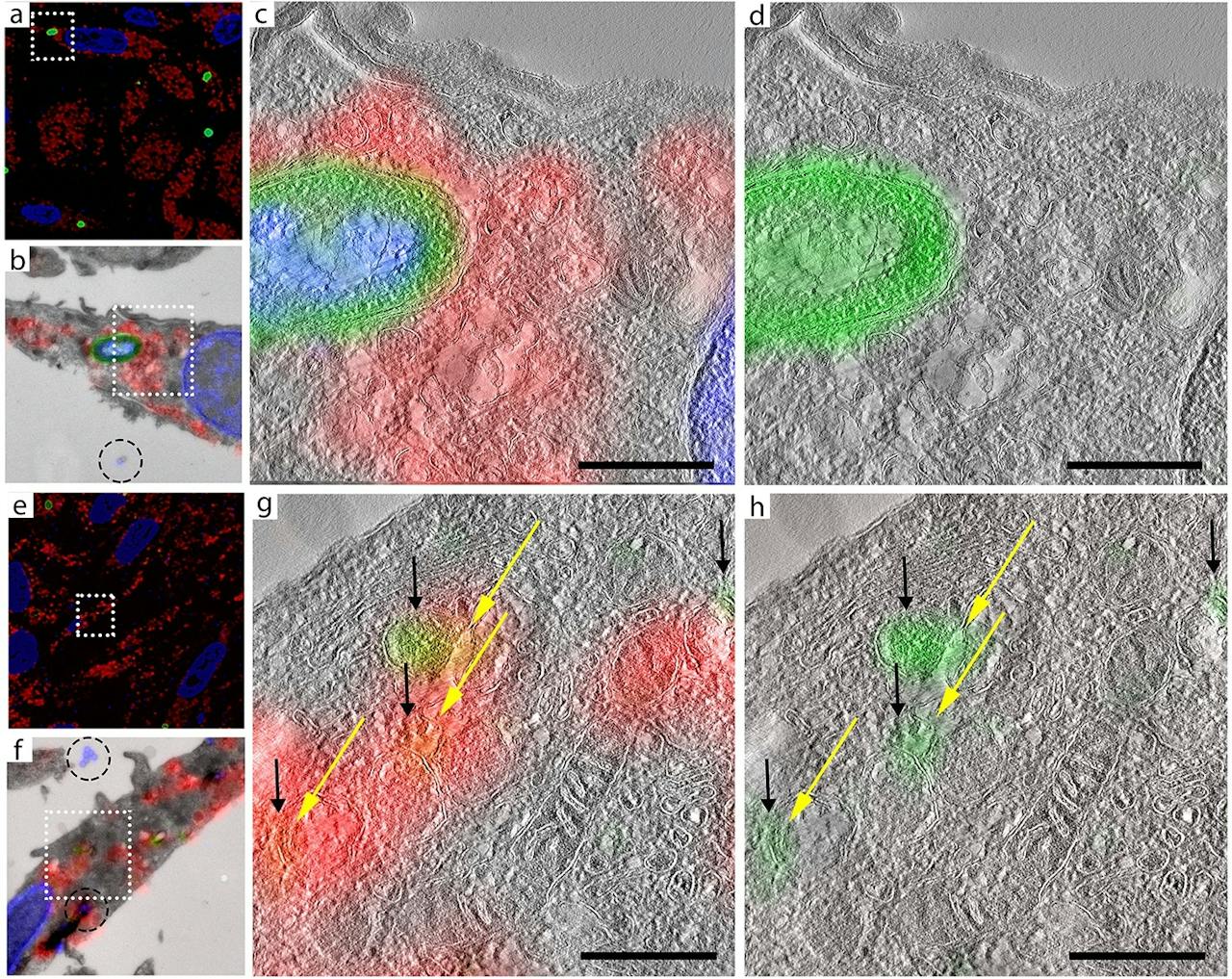
Opnames A-D zijn gemaakt kort na het samenvoegen van de bacteriën en de immuuncellen. A: afbeelding gemaakt met de fluorescentiemicroscoop. De hele bacterie is nog zichtbaar (groen). B: Detail uit afbeelding A gecorreleerd met een elektronenmicroscopiebeeld. Nu is de cellulaire omgeving van de bacterie (groene ovaal) te zien. C en D: detailweergaves van B. De intacte bacterie is duidelijk herkenbaar in het groen. Blauw markeert DNA, van zowel de immuuncel als de bacterie. Rood markeert structuren die betrokken zijn bij de afbraak van bacteriën. Opnames E-H zijn vergelijkbaar met A-D, maar dan genomen op een later tijdstip. E: opname met de fluorescentiemicroscoop, de bacterie (groen) is duidelijk in omvang afgenomen. F: correlatie van detail uit E met een elektronenmicroscopische opname. G en H: detailweergaven van F, de groene fluorescentie afkomstig van de bacteriële eiwitten is nu te zien in zogeheten ‘vesiculaire’ structuren of blaasjes (zwarte pijlen). Het blauwe signaal van het DNA van de bacterie is niet langer zichtbaar, wat erop wijst dat de bacterie niet langer intact is. We zien hier daadwerkelijk afgebroken bacteriën. Bovendien zien we dat deze afbraakproducten zich vlakbij onderdelen (gele pijlen) bevinden die betrokken zijn bij de afbraak van bacteriën.
Daphne van Elsland/Universiteit Leiden CC1.0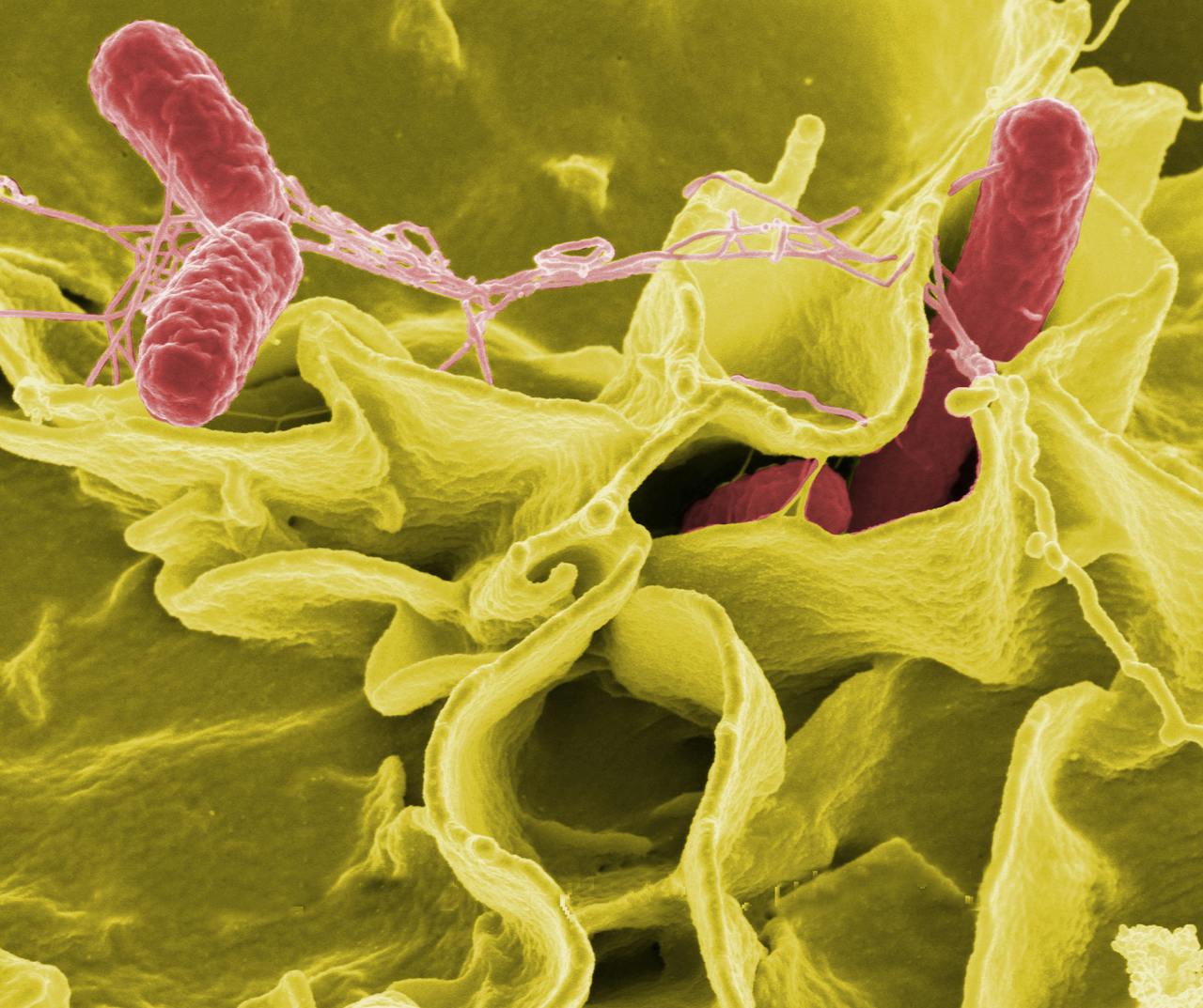
Salmonellabacteriën (rood) dringen een immuuncel (geel) binnen.
NIAID via Wikimedia Commons CC BY 2.0Salmonella
Ze gaat zich nu meer richten op het afbraakproces zelf. “Ik ga bijvoorbeeld werken met andere niet-natuurlijke aminozuren en niet alleen eiwitten labellen, maar ook suikers. Bacteriën hebben aan hun buitenkant allemaal verschillende suikermoleculen en die zijn belangrijk voor de herkenning door immuuncellen.”
Door daar een label aan te koppelen, hoopt Van Elsland te ontdekken waarom sommige bacteriën, zoals Salmonella en Mycobacterium tuberculosis (de veroorzaker van tuberculose), niet worden afgebroken door immuuncellen en ongehinderd kunnen voortbestaan.