Steeds meer bacteriën bouwen resistentie op tegen antibiotica, wat leidt tot schaarsheid van deze bacteriedodende stofjes. Wetenschappers van de universiteit Leiden vonden met kunstmatige intelligentie 42 nieuwe, potentiële antibiotica in een familie van bodembacteriën.
Een simpele snee in je hand, gevolgd voor een bacterie-infectie kon in de zeventiende eeuw je einde betekenen. Door gebrek aan hygiëne en medicijnen konden gevaarlijke ziekteverwekkers via een snee eenvoudig je lichaam binnenkruipen, gifstoffen maken, zich vermenigvuldigen en enorme ontstekingsreacties veroorzaken met de dood als gevolg. Tegenwoordig hebben we schoon drinkwater, ontsmettingsmiddelen en betere hygiëne, waardoor minder mensen in de bloei van hun leven sterven aan een ontsteking. Bovendien kunnen we infecties nu bestrijden met een antibioticakuur.
Tegenwoordig bestrijden we infecties zoals longontstekingen en bloedvergiftiging met een simpele antibioticakuur.
Pixabay, Alina Kuptsova via CC0Eigenschappen vergelijken
De ontdekking van antibiotica was een enorme sprong vooruit in de geneeskunde. Maar een nieuw gevaar ligt op de loer. Vanwege veelvoudig gebruik van antibiotica in de zorg en veehouderij komen bacteriën regelmatig in aanraking met deze voor hen giftige stofjes en ontwikkelen ze er resistentie tegen. De klassieke manier van nieuwe antibiotica opsporen, met kweekjes van schimmels en bacteriën, raakt uitgeput. Daarom pakte de Leidse promovendus Alexander Kloosterman het anders aan. Hij gebruikte kunstmatige intelligentie om nieuwe bacteriedodende stofjes op te diepen.
Kloosterman en zijn collega’s richtten zich op een groep bodembacteriën die van nature antibiotica maken. Die stofjes bestudeerden de biotechnologen op moleculair niveau. Ze onderzochten welke bouwstenen (aminozuren) deze antibiotica bestaan en welke eigenschappen ze hebben. Sommige bouwstenen hebben een kleine positieve of juist negatieve lading, andere houden van water of zijn waterafstotend.
Om nieuwe antibiotica op te sporen, scanden de wetenschappers het DNA van bodembacteriën. Ze zochten naar eiwitten waarvan de eigenschappen lijken op bestaande antibiotica. “Dat is te ingewikkeld om met de hand te doen”, vertelt Kloosterman. Dus schakelden de onderzoekers de hulp van kunstmatige intelligentie in. In het DNA van honderden bodembacteriën spoorde de computer een flinke hoeveelheid potentiële antibiotica op.
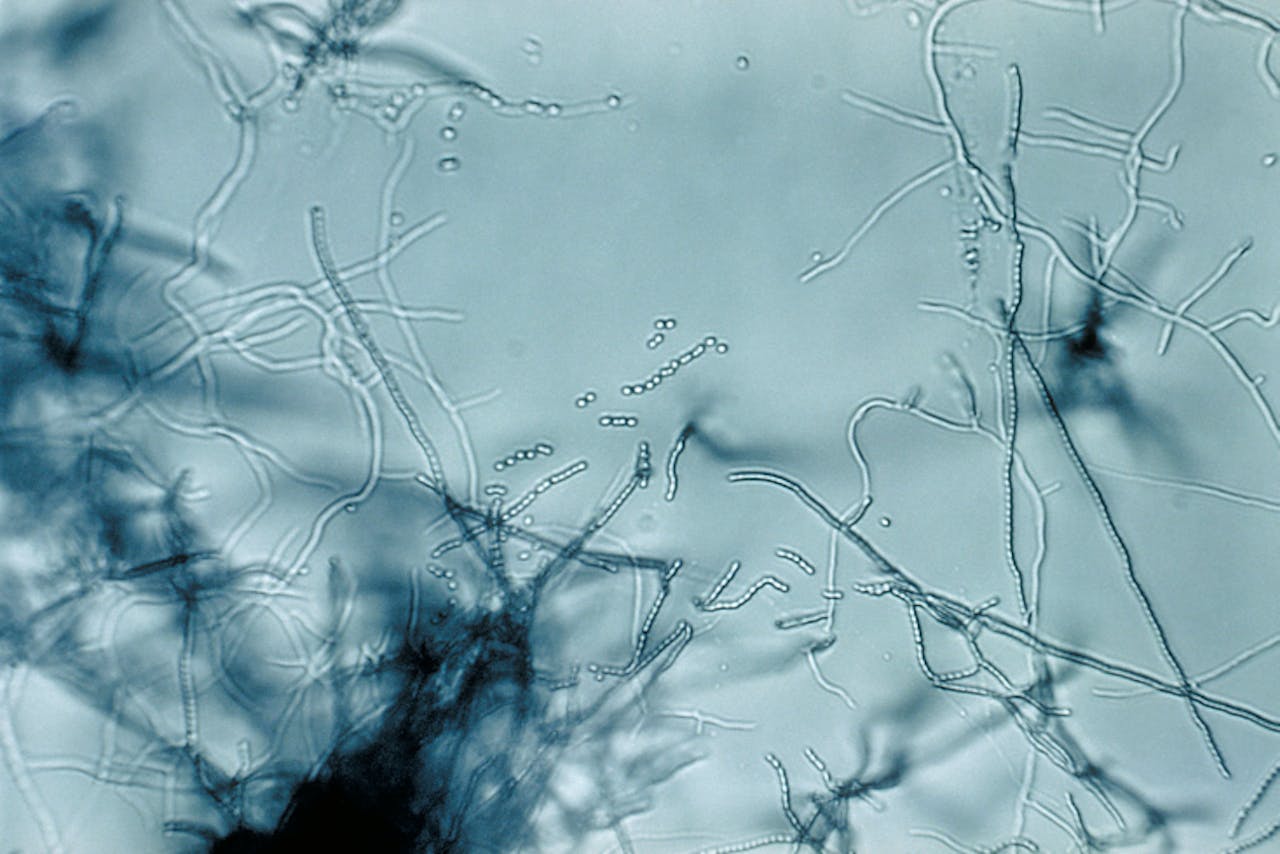
Een microscopische afbeelding van bodembacteriën (streptomyceten). In dit soort bacteriën vonden de Leidse onderzoekers 42 nieuwe, potentiële antibiotica.
Wikimedia Commons, public domain“Daar zaten alsnog veel andere stofjes tussen”, zegt Kloosterman. Daarom keken de wetenschappers naar de omliggende genen. “Rondom een gen dat de blauwdruk bevat van het antibioticum, liggen genen die extra informatie geven over bijvoorbeeld de aanmaak en het vervoer van dat stofje”, licht Kloosterman toe. Werd het gen van een potentieel antibioticum dus niet omringd door dit soort genen, dan verdween die uit de selectie.
Nieuw antibioticum pristinine
De uiteindelijke lijst met 42 nieuwe potentiële antibiotica publiceerden Kloosterman en zijn begeleiders Gilles van Wezel en Marnix Medema in het wetenschappelijk tijdschrift PLoS Biology. Eigenlijk zijn het er nog meer, want ieder van die 42 heeft een aantal varianten waarbij de chemische samenstelling net iets anders is. Dat kan betekenen dat de ene variant wel werkt, en de ander niet. Of dat beide varianten bacteriedodend zijn, maar de een beter werkt dan de ander.
Van de nieuw ontdekte stoffen maakte Kloosterman er zelf één in het laboratorium. Dat deed hij door het gen voor dat stofje, genaamd pristinine, en de omliggende genen in te schakelen in de bodembacterie. “Helaas maakte de bacterie weinig van het stofje”, zegt Kloosterman. Daardoor konden ze pristinine nog niet loslaten op andere bacteriën om de bacteriedodende werking te bevestigen. Wel ontrafelde Kloosterman de chemische structuur van het antibioticum. Die leek erg op die van een eerder ontdekt bacteriedodend stofje. Daarom vermoeden de Leidse wetenschappers dat ook pristinine bacteriën doodt.
Testen in het lab
De lijst met antibiotica en het computerprogramma zijn nu openbaar. “Collega-wetenschappers mogen de potentiële antibiotica testen in hun laboratorium”, aldus Kloosterman. Op die manier hopen ze er snel achter te komen welke stofjes artsen in de toekomst kunnen gebruiken om infecties te behandelen.
Niet alleen in Leiden sporen onderzoekers nieuwe antibiotica op met kunstmatige intelligentie. Verschillende wetenschappers wereldwijd komen met eenzelfde soort methode. “Omdat iedereen op een andere manier programmeert, verschillen de uitkomsten en het is interessant om die met elkaar te vergelijken”, zegt Kloosterman. “Misschien is het ene programma wel helemaal fout en het ander bijna helemaal goed. Of heeft ieder programma een klein deel goed.”
Welk programma’s juist zijn en welke stofjes daadwerkelijk bacteriedodend zijn, ontdekken onderzoekers alleen maar door het laboratorium in te duiken. In de komende jaren testen wetenschappers dus potentiële antibiotica, strepen ze enkele van de lijst af en – zo hopen de Leidse biotechnologen – ontdekken ze een hoop nieuwe antibiotica. Die moeten ons beschermen tegen infecties zodat de gezondheidszorg niet tegen dezelfde problemen aanloopt als in de zeventiende eeuw.

